そもそもSU剤って
そもそもSU剤は,どうやって膵臓からインスリンを『絞り出して』いるのでしょうか?
血糖値が高くなると 膵臓β細胞からインスリンが分泌される仕組みは,教科書では下図のように説明されています.
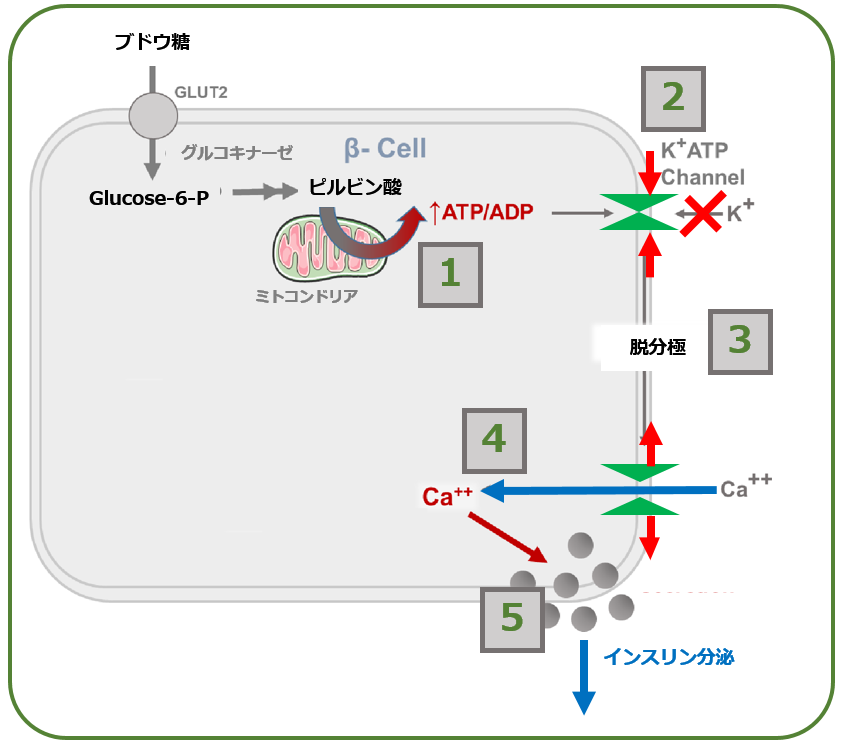
[1] 血糖値が高くなると,ブドウ糖はグルコキナーゼによってβ細胞内に取り込まれて,ミトコンドリア内でATPの産生が増加します.
[2] β細胞では「ATPが多くなった」=「血糖値が上がった」と解釈して,細胞膜のK+_ATPチャネル(Kir)★が閉じます.
[3] すると細胞膜の両側の電位差が小さくなる(=脱分極)ので,
[4] Ca++チャネルが開き,細胞内のCa++濃度が上がります.
[5]この刺激により,β細胞からインスリンがドバーッと(音はしませんがw)細胞外に分泌されます.
★『Kir』= Inward Rectifying Potassium channel :内向き整流カリウムチャネル=細胞膜の外から内側だけに一方通行のカリウムイオン透過チャネル
実によくできてますね.
そして,SU剤によってインスリン分泌が行われる仕組みはこうです.
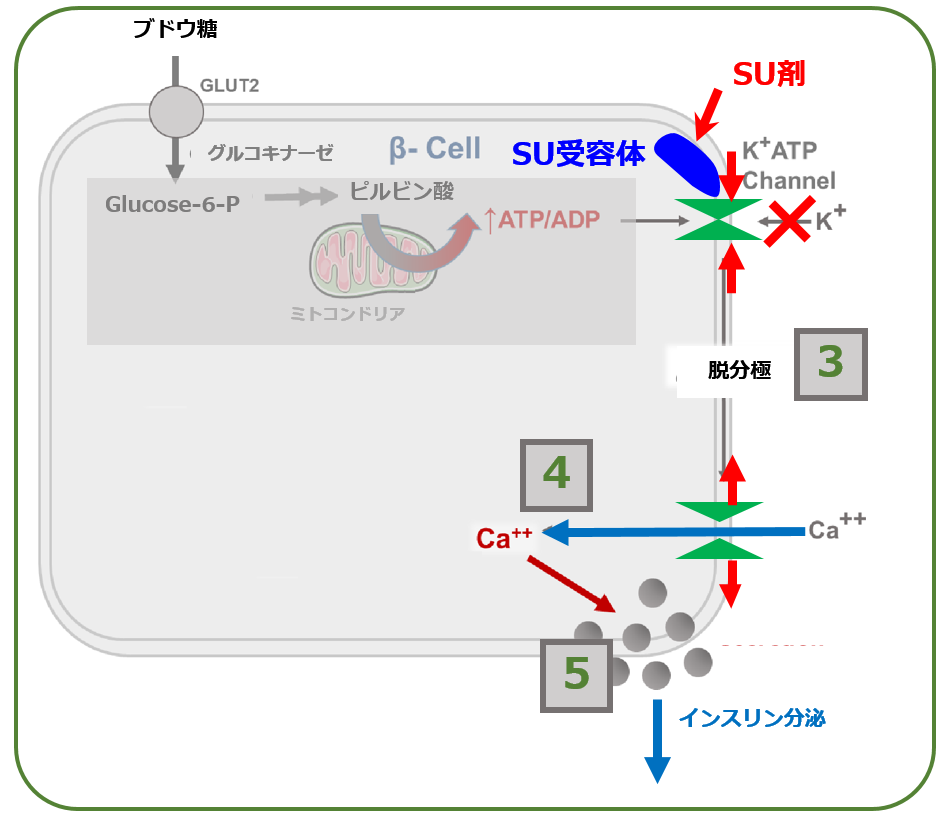
前記の K+ATPチャネルの周りには,スルホニルウレア受容体[SU受容体]があり,ここにスルホニルウレア薬物(SU剤)が到達すると上記と同じことが起こるのです.ATP濃度が高まっていなくても,すなわち血糖値が高くなっていなくても,K+_ATPチャネルは強制的に閉じられ,以下 最初の図と同じ手順でインスリンが分泌されます.
ええっ? スルホニルウレア受容体??
しかし,この話 おかしくはありませんか? ぞるばだけかもしれませんが,この『スルホニルウレア受容体(SU受容体)』という名前を初めて聞いた時,非常に不思議に思いました.
なぜなら SU剤は人間が最近発明したものであって,自然界には存在しないものだからです.人体内に存在するはずのないSU剤のために,SU剤専用の受容体が人体に予め用意されているとは 不思議な話です.
そこで もう少し詳しくこのSU受容体を調べてみました.
SU受容体とは,K+_ATPチャネルの周辺部を構成するタンパク質です. K+_ATPチャネル全体の構造はこうなっています.
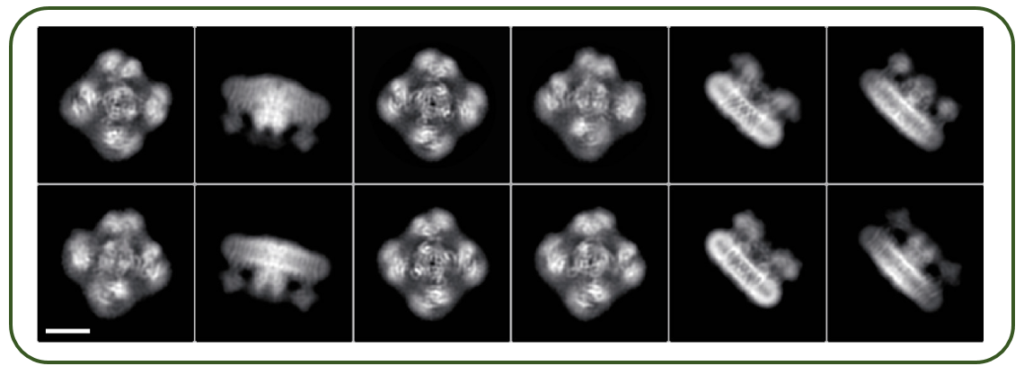
極低温電子顕微鏡(Cryo EM )を使って,直接 K+_ATPチャネルをいろいろな方向から観察したものです.すごいですね. 現在では 直接こうやって観察できるのですから.
ご覧の通り,K+_ATPチャネルは 4つの部品が集まったような形をしています.
このタンパク質の構造解析によれば;
横からみた場合;
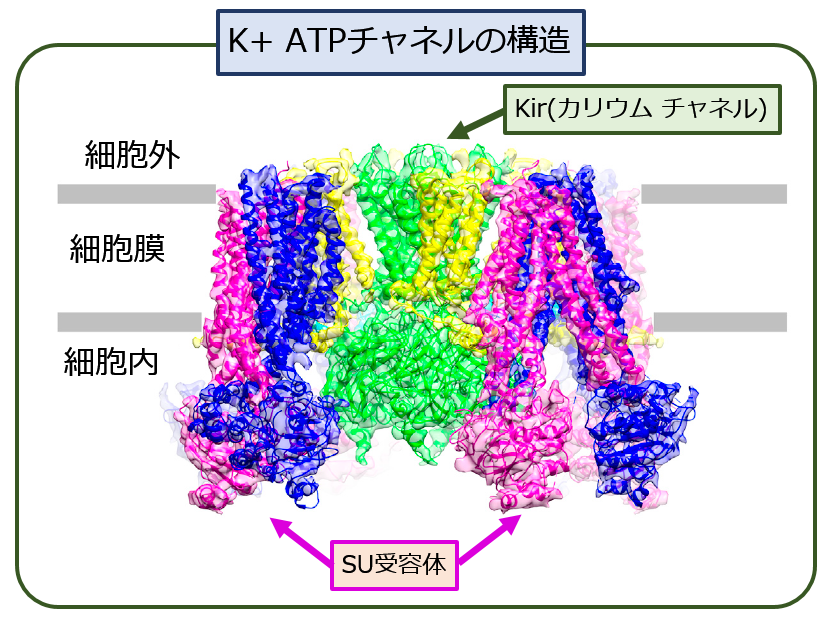
真上から見るとこうです.
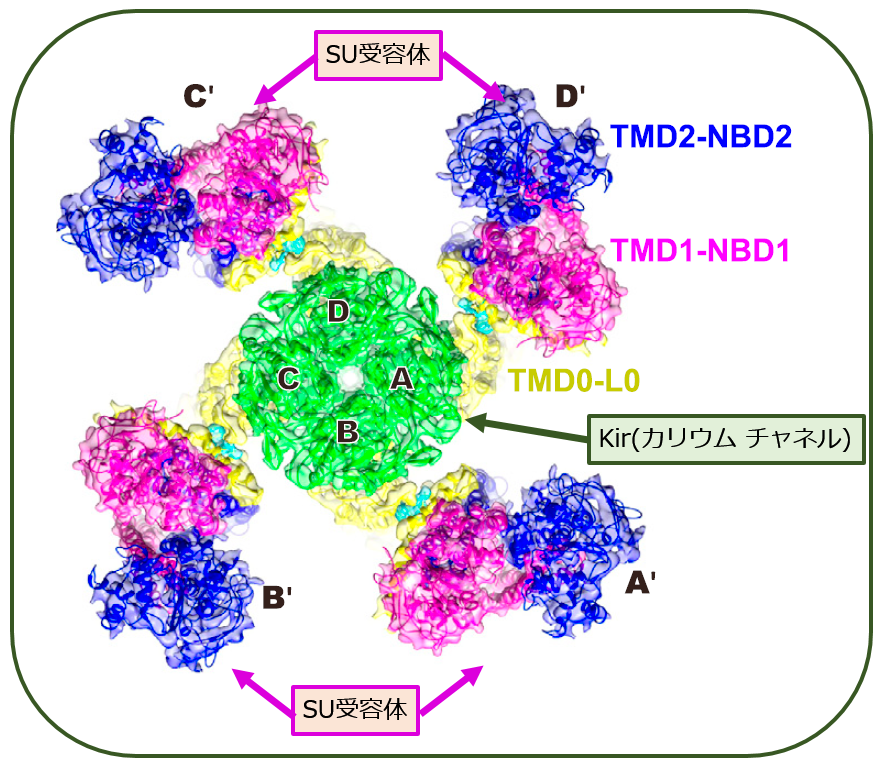
まるでチアリーダーのように華やかですが,実物には もちろんこんな色は付いていません.
中心にあるKir(内向き整流カリウムチャネル)は,細胞外からK+,つまりカリウムイオンだけを一方通行的に取り込む『孔』(チャネル)です.この記事 にも書いたように,イオンを取り込むチャネルは非常に選択性が高く,たとえばナトリウムイオン(Na+)は,カリウムイオンより直径は小さいのに,このK+_ATPチャネルを通過できません.したがって,このK+_ATPチャネルの中心にある『孔』は,単なるトンネルではなく,非常にややこしい構造をしています. しかも,その『孔』の開閉は,厳密に細胞内部のATPの濃度によって制御されています. 細胞内のATP濃度が低い時=人体の血糖値が低い時には,この『孔』は開いており,細胞内のATP濃度が高くなると=人体の血糖値が高くなると,この『孔』は開じます.
中心のKirを 取り囲む 4つのボンボンがSU受容体です.
では,K+_ATPチャネルは,どうやって細胞内のATP濃度が高い/低いを知るのでしょうか?
それこそがスルホニルウレア受容体(SU受容体)の役目なのです.
スルホニルウレア受容体ではなかった
一言で言えば,スルホニルウレア受容体(SU受容体)は スルホニルウレアを取り込むために存在しているのではありません. こういうことです.
K+_ATPチャネルは,常に細胞内のATP濃度に応じて,K+を通過させる『孔』を閉じたり開いたりしています. その仕組みは;
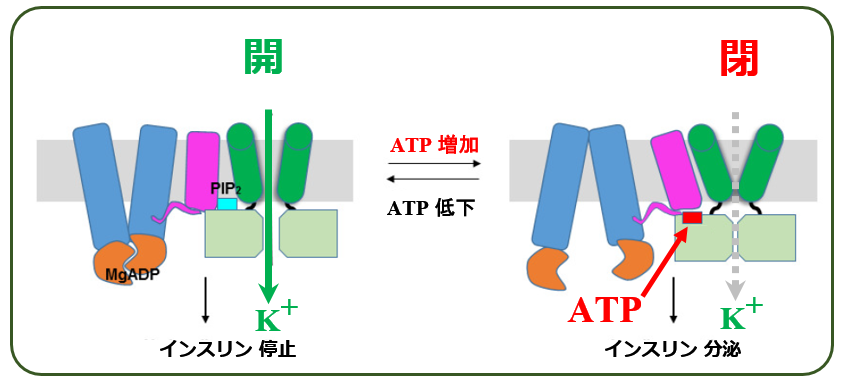
上図の通り,SU受容体には,その底面(細胞の内側を向いています)にATPのセンサーがあり,ここにATPが取りつくと,構造が変化します. するとSU受容体に結合しているKirも,それに引っ張られてねじれる様に構造変化して,その中心が細くなります[図 右].つまりこれで『孔』が閉じるのです. ですから SU受容体は,K+_ATPチャネル に附属して 常に現在のATP濃度を知らせているセンサー担当なのです.そして これがSU受容体の本来の役目なのです.
では,どうしてSU受容体はそう呼ばれるようになったのでしょうか? それは上図で SU受容体にATPが取りつくと,SU受容体とKirの間に少し【隙間】(図左の「PIP2」の左)が変化することに関係しています.この【隙間】が 偶然にもスルホニルウレアが 割り込んでおさまるのにちょうどいい大きさと形をしていたのです. なので,スルホニルウレア(SU)が細胞の付近に存在すると,

このように 勝手にこの隙間を広げて入り込んでしまい,結果としてKirは 『あ, 今 ATPが高いのだな』と勘違いして,『孔』が閉じてしまいます.
もちろんこれは 血糖値の高い低いにはまったく無関係に,ただSU剤が存在するだけで 必ず起こりますから,結果として;
スルホニルウレア(SU剤)は,血糖値と関係なく インスリンを分泌させる
という作用を示すのです. ですから 『スルホニルウレア受容体(SU受容体)』という名称を付けられてしまったことは,本人にとっては まことに不本意な話で,『俺はそんな目的のために ここにいるんじゃないっ!』と叫びたいでしょうね.
こんな 名前はいやだっ!!

正確には,『スルホニルウレア受容体(SU受容体)』とは,『細胞内のATP濃度を適時に知らせるという本来の役目を,SU剤によりブロックされてしまうもの』というべきでしょう.
[続く]

コメント
>俺はそんな目的のために ここにいるんじゃないっ!
あはは。
>『スルホニルウレア受容体(SU受容体)』という名前を初めて聞いた時,非常に不思議
わたしも、まさにそう思っていました。
SU受容体という名称はSU薬の作用を説明する分には便利だけど、生体内での本来の作用が分かりにくくなってしまいますよね。
SUR1の正式な名称はABCC8、ATP-binding cassette, subfamily C, member 8 です。よく保存されたATP結合ドメインをもつ膜タンパク質ファミリーのメンバーであることを示しています。
当初はSU薬が結合して作用を示すタンパク質をコードした遺伝子としてクローニングされたのでSU受容体と名付けられたけれど、その後に機能面、配列面から整理されて、正式な遺伝子名が与えられたわけです。
Kir6.2も同じで、正式な名称はKCNJ11、potassium channel, inwardly rectifying, subfamily J, member 11 ですね。
遺伝子名が変更になるのはよくあることだけど、新しい名前は分類的には便利な一方で、無機質で取っつきにくいです。なので、SUR1が「オイラの本当の名前はABCC8だい!」と憤っているかどうかは難しいところですかね〜w
ちなみに、SURと同じようにmTORもmechanistic target of rapamycin (あるいはmammalian target of rapamycin)と、「ラパマイシンの標的分子」という意味です。こちらはmTORが正式名称として使われています。mTORこそ「こんな名前は嫌だー!」と思っているかも?
>SU受容体は,K+_ATPチャネル に附属して
わたしの理解では、KATPチャネルはKir6.2サブユニットとSUR1サブユニットから構成されているって感じですかね。Kir6.2とSUR1が1対1で対になっていて、それが4つ集まった8量体として機能している。SUR1はKATPチャネルの制御サブユニットで、Kir6.2はポアを形成するサブユニットである、と。
興味深いのは、Kir6.2遺伝子とSUR遺伝子が並んでコードされていることです。
お隣さん同士で協力し合ってるんですね〜
>わたしも、まさにそう思っていました
やっぱり おかしいんですよね. シロウトは こういうところで「ん?」とつまずいてしまうと,その疑問解明に ものすごく手間取るのですよ. 『責任者,出てこいっ!』(←[C]人生幸朗 ← 古いっ! )
https://www.youtube.com/watch?v=FfcQhfLi37w
そういえば やはりシロウト,K+の移動方向を間違えてましたね. 後で図を訂正しておきます.